中国人民大学,统计学院,北京 100872
多种环境因素(包括辐射,热量和氧自由基)引起的DNA损伤可在DNA复制过程中诱导突变。研究表明,基于原子力显微镜(AFM)单分子操纵技术的机械力可诱导DNA损伤及在DNA复制过程中诱导突变。然而,并没有进一步证明其结果有没有显著统计学意义。本文旨在用卡方检验方法(Chi-square)来比较三种DNA样品(纳米操纵DNA(n=22),拉伸DNA(n=223),稀释DNA(n=180))中的DNA突变比率。从而得出机械力诱导DNA突变比率和拉伸DNA、稀释DNA中的突变比率相比具有显著统计学意义。
关键词:卡方检验(Chi-square test),DNA突变,纳米操纵
前言
DNA 突变(DNA mutation)是DNA结构中碱基所发生的改变。DNA突变是生命进化的动力。复制过程中聚合酶的不准确性是导致DNA突变的重要原因。其他原因包括遗传物质的物理或化学伤害[1-3],如辐射,热量和氧自由基。随着分子生物学和诱变技术的进步,越来越多方法都能诱发遗传物质的变化[4]并定性检测基因变化[5, 6]。
迄今为止,对于这种机械力是否会在操纵过程中引起DNA损伤尚知之甚少。在体内条件下,DNA分子在许多生物学过程中都受到机械力的作用[7],DNA转录,基因表达和DNA复制。例如,在复制过程中,必须强制解开由DNA解旋酶产生的亲本DNA链。尚不清楚机械力是否会导致DNA损伤和突变。因此,重要的是在一定的力存在下测量DNA的损伤和突变显得尤为重要。
光镊和扫描探针显微镜可以操纵单个DNA分子或蛋白质[8]。对单个DNA分子的广泛研究集中于其机械性能[7]。原子力显微镜(AFM)不仅可以在单分子水平上显示和操纵衬底上的天然生物样品[9, 10],而且可以精确地控制AFM对生物样品施加的机械力 [11, 12]。在之前的研究中,我们研究了机械力诱导的DNA突变[13]基于原子力显微镜(AFM)的单分子操作,单分子聚合酶链反应(SM-PCR)和Sanger测序的结合 [14-16]。在本文中,我们旨在研究使用卡方检验方法得出P值来评估基于AFM的单分子操纵DNA与未操纵的DNA相比DNA的突变率是否会有不同。
实验材料与方法
DNA提取
569bp DNA 自pBR322 (TaKaRa Bio. Inc., Dalian, China)扩增(3,632th to 4,200th),聚合酶为Ex Taq DNA polymerase Hotstart (TaKaRa Bio. Inc.),随后纯化保存在TE缓冲液中。
PCR 扩增
DNA片段引物:
(P1: 5’-GTCGTTTGGTATGGCTTCA-3’)
(P2: 5’-GACAATAACCCTGATAAATGCT-3’)。由上海生工生物工程有限公司合成。 “分子梳”方法[13, 16]将DNA制于3-氨丙基三乙氧基硅烷( (3-aminopropyl)-triethoxysilane,APTES)修饰的云母衬底上[17]。DNA的成像和操作是通过多模AFM和硅悬臂在空气中进行的。为了进行操作,将AFM尖端向下放到表面并扫描目标DNA分子。然后,通过原子力显微镜尖端分离云母上的目标DNA分子。如图1所示,将带有AFM尖端的分离的DNA片段(569 bp)转移到灭菌的PCR管中,并通过单分子PCR进行扩增。
在对照组中,将作为主要对照组的稀释DNA用作PCR模板。此外,首先将DNA分子在APTES-云母上拉伸[17,18],然后用次要对照组Milli-Q水洗脱。稀释洗脱的DNA,并将其用作PCR模板,如图1所示。
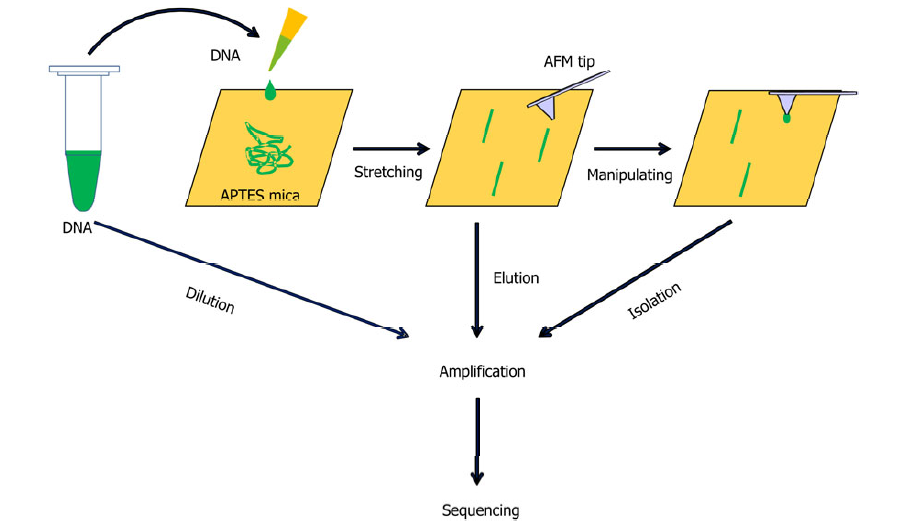
图1 不同组中DNA的扩增和测序,包括稀释,拉伸和操纵。该实验中使用的DNA分子的长度为569 bp [13]
统计分析
卡方检验用于评估操纵与DNA突变之间是否存在关联 [19]。皮尔逊(Pearson)卡方分布的重要性在于,统计学家可以使用不依赖于正态分布的统计方法来解释发现。计算卡方统计量的公式为:
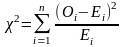
其中,O代表观测频率,E代表期望频率。从观察到的计数中减去期望的计数以找到差异。然后计算差值的平方以消除负值。然后,将差的平方除以期望值以归一化。它们前面的sigma符号表示我们为每个单元格加和计算出这些值。由于DNA样品是随机收集的,所以该统计方法适用。所研究的变量是分类变量,在分类变量的每个水平上,每类分类变量的期望频率计数至少为5。 零假设是H0,它表示稀释DNA中的突变比例与操纵DNA中的突变比例相同。备择假设是Ha:它表示稀释DNA中的突变比例与操纵DNA中的突变比例不同.对于此分析,我们选择显著性水平α为0.05。卡方检验第一步是建立一个 2 × 2 列联表 (表1)。 用于从特定单元格的观察计数中计算期望计数的通用公式为 [(行总和×列总和)/总人数(表2)。 首先,计算自由度(df)。当一个样品与另一个样品进行比较时,自由度 = (列数 − 1)× (行数 − 1)。因此,此试验中的自由度为 1. 基于卡方检验与样本数据的均一性,我们计算了df,期望频率计数和卡方检验统计量。并基于卡方统计量和df计算P值。
表1:2×2 列联表的一般符号
| 实验组DNA | 对照组DNA | 合计 |
突变 | a | b | a+b |
无突变 | c | c | c+d |
合计 | a+c | b+d | a+b+c+d=n |
表2:2×2 列联表中的期望频数的计算
| 实验组DNA | 对照组 DNA |
突变 | (a+b) (a+c)/n | (a+b) (b+d)/n |
无突变 | (a+c) (c+d)/n | (c+d) (b+d)/n |
实验结果
我们共收集了187个拾取了DNA片段的AFM探针并进行PCR。由于单分子PCR效率低下,仅有22个样品被成功扩增并测序。同时,577份DNA样品(281份稀释DNA;296 份拉伸DNA) 作为对照组,并获得403份PCR和测序结果 (其中,180 份稀释DNA,223 份拉伸DNA)。
根据实验结果,我们发现基于AFM的单分子操纵导致了DNA突变率增加约70%。在使用稀释和拉伸DNA进行对照实验的情况下,突变率分别约为30%。
表3中给出了用于计算卡方检验的操纵与DNA突变之间的关联的数据。操纵DNA为实验组稀释DNA为对照组。卡方检验统计量可以有上述公式得出。参考表 4 和表5 进行计算。根据得出的卡方统计量和自由度,算出的P值为0.0002。统计检验结果在0.05水平上拒绝原假设即两比率相等。从而得出AFM操纵的DNA发生突变的概率与稀释DNA突变概率相比具有显著统计学意义。因此,AFM操纵可以有效提高DNA的突变率。这两个比例之间的差异的置信区间为(0.1983, 0.6451)。 根据以上统计分析结果我们得出AFM操作与DNA突变之间存在关联。
表3:用于计算卡方统计量的观察频数 (操纵 DNA vs 稀释 DNA)
| 操纵 DNA | 稀释 DNA | 合计 |
突变 | 16 | 55 | 71 |
无突变 | 6 | 125 | 131 |
合计 | 22 | 180 | 202 |
表 4:期望频数 (操纵 DNA vs 稀释 DNA)
| 操纵 DNA | 稀释 DNA |
突变 | (71×22) / 202 = 7.732673 | (71 × 180) / 202 = 63.26733 |
无突变 | (131 × 22) / 202 = 14.26733 | (131 ×180) / 202 = 116.7327 |
表5:计算卡方统计量的汇总数据
观察频数( | 期望频数 ( | | |
16 | 7.732673 | 68.3487 | 8.8390 |
55 | 63.26733 | 68.3487 | 1.0803 |
6 | 14.26733 | 68.3487 | 4.7906 |
125 | 116.7327 | 68.3487 | 0.5855 |
| | | 13.501 |
当然,我们也可以用R来计算卡方统计量:
M <- as.table(rbind(c(16, 55), c(6, 125)))
dimnames(M) <- list(outcome = c("Mutations", "Non-Mutations"),
Treatment = c("Manipulated DNA","Diluted DNA"))
(Xsq <- chisq.test(M))
优势比 (OR) 计算如下:
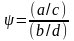
OR值为 6.06 表明AFM操纵的DNA突变率比稀释DNA高约6倍。 我们知道AFM操纵DNA的过程可以大致分为两个步骤:第一步,通过“分子梳”技术将稀释后的DNA分子拉伸到APTES修饰的云母[17]表面上[13, 16];第二步,AFM对DNA进行成像和操纵。那么,是哪一步诱导了DNA突变呢?
比较拉伸DNA和稀释DNA的突变率卡方统计量为0.477如表 6所示, df = 1。根据卡方统计量和自由度得出P 值为 0.4898。因此,不能拒绝原假设,在0.05水平上两样本的突变率是一样的。即拉伸DNA和稀释DNA的突变率的不同没有显著统计学意义。
表6:用于计算卡方统计量的观察频数 (拉伸DNA vs 稀释 DNA)
| 拉伸DNA | 稀释 DNA | 合计 |
突变 | 62 | 55 | 117 |
无突变 | 168 | 125 | 293 |
合计 | 223 | 180 | 410 |
比较操纵DNA和拉伸DNA的突变率卡方统计值如表 7所示,统计量为 17.599 (df = 1)。根据卡方统计量和自由度得出P 值为 0.0001。由于P>α=0.05。拒绝原假设,结果表明操纵DNA的突变率与拉伸DNA突变率之间存在显著差异。操纵处理对DNA突变有效,在实验中起着最重要的作用。
表7:用于计算卡方统计量的观察频数 (操纵DNA vs 拉伸 DNA)
| 操纵DNA | 拉伸DNA | 合计 |
突变 | 16 | 62 | 78 |
无突变 | 6 | 168 | 174 |
合计 | 22 | 223 | 245 |
讨论
在该实验中,DNA突变的频率在操纵的DNA组中明显高于稀释的DNA或拉伸的DNA组。经稀释和拉伸处理的DNA样品之间未发现显着差异。突变率的增加表明,在操作过程中,机械力可能会对DNA分子造成某种损害,并导致PCR扩增后发生突变。此外,我们对DNA突变率拟合logistic回归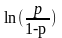
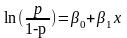
此公式中,p为 DNA突变率Y, p=(Y=1); p/(1-p) 即比值; 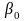 ,
,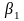 是根据数据最大似然估计估计的回归系数。
是根据数据最大似然估计估计的回归系数。
回归结果如下:
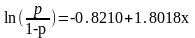

基于这些结果,显着性水平为0.05时,操纵DNA与稀释DNA相比具有显着区别。然后我们得出结论,DNA操纵与DNA突变可能性的变化有关。这表明,如果DNA受AFM操纵,则DNA突变比稀释的DNA高约6.0倍。
在操纵DNA样本中的预测突变率为72.7%稀释DNA样本为30.6%。
并且我们得出了操纵DNA突变率与稀释DNA突变率相比较的的卡方检验的p值作为主要分析终点,操纵DNA突变率与拉伸DNA突变率相比较的的卡方检验的p值作为次要分析终点,同时得出了拉伸DNA突变率与稀释DNA突变率相比较的的卡方检验的p值。因此,由于我们做了三次比较,有可能增加I型错误。所以我们将3组一起在显著性水平为0.05的基础上做卡方检验。
我们可以用R来计算:
M <- as.table(rbind(c(16, 62,55), c(6, 168,125)))
dimnames(M) <- list(outcome = c("Mutations", "Non-Mutations"),
Treatment = c("Manipulated DNA", "Stretched DNA","Diluted DNA"
))
(Xsq <- chisq.test(M))
比较操纵DNA,拉伸DNA, 稀释DNA突变率的卡方统计量如表8所示,P值为5.147e-05。从而拒绝原假设,表明三种DNA样品突变率在0.05水平上有显著差异。也就是说,三组DNA样品中有一组或两组对DNA突变有效。
根据分析结果,操纵处理对DNA突变有效,在实验中起着最重要的作用。
表8:用于计算卡方统计量的观察频数 (操纵DNA vs 拉伸 DNA vs 稀释 DNA)
| 操纵DNA | 拉伸DNA | 稀释DNA | 合计 |
突变 | 16 | 62 | 55 | 133 |
无突变 | 6 | 168 | 125 | 299 |
合计 | 22 | 223 | 180 | 432 |
结论
我们给出了一种统计学方法-卡方检验用于比较两个(或多个)独立组之间的分类响应计数或计数。
本文用卡方检验方法来检验DNA突变率在纳米操纵DNA,拉伸DNA,稀释DNA中是否存在显著差异,得出在0.05水平上纳米机械力操纵DNA可以增加DNA突变率。
通过分析每个过程中的突变结果,探索由机械力引起的非随机突变的可能性将非常有趣。
参考文献
1. Watson, J., Molecular Biology of the Gene: United States Edition 6 Book paper plus CD ROM - 1 ISBN.
2. Kunkel, T.A., The high cost of living. American Association for Cancer Research Special Conference: endogenous sources of mutations, Fort Myers, Florida, USA, 11-15 November 1998. Trends Genet, 1999. 15(3): p. 93-4.
3. Lindahl, T., Instability and decay of the primary structure of DNA. Nature, 1993. 362(6422): p. 709-15.
4. Wood, M.L., et al., Mechanistic studies of ionizing radiation and oxidative mutagenesis: genetic effects of a single 8-hydroxyguanine (7-hydro-8-oxoguanine) residue inserted at a unique site in a viral genome. Biochemistry, 1990. 29(30): p. 7024-32.
5. Muyzer, G., DGGE/TGGE a method for identifying genes from natural ecosystems. Curr Opin Microbiol, 1999. 2(3): p. 317-22.
6. Li, J., et al., Replacing PCR with COLD-PCR enriches variant DNA sequences and redefines the sensitivity of genetic testing. Nat Med, 2008. 14(5): p. 579-84.
7. Bustamante, C., Z. Bryant, and S.B. Smith, Ten years of tension: single-molecule DNA mechanics. Nature, 2003. 421(6921): p. 423-7.
8. Merkel, R., et al., Energy landscapes of receptor-ligand bonds explored with dynamic force spectroscopy. Nature, 1999. 397(6714): p. 50-3.
Biological atomic force microscopy: from microns to nanometers and beyond. Annu Rev Cell Dev Biol, 1995. 11: p. 241-65.
10. Drake, B., et al., Imaging crystals, polymers, and processes in water with the atomic force microscope. Science, 1989. 243(4898): p. 1586-9.
11. Hu, J., et al., Artificial DNA patterns by mechanical nanomanipulation. Nano Letters, 2008. 2(1): p. 55-57.
12. J., et al., Nanomanipulation of single DNA molecules and its applications. Surface & Interface Analysis, 2004.
13. Zhang C, D.N., Bin D, et al, Nuclear Science and Techniques, 2013: p. 5331: 1518- 1523.
14. Lü, J.H., et al., Positioning isolation and biochemical analysis of single DNA molecules based on nanomanipulation and single-molecule PCR. J Am Chem Soc, 2004. 126(36): p. 11136-7.
15. An, H., et al., Single-base resolution and long-coverage sequencing based on single-molecule nanomanipulation. Nanotechnology, 2007.
16. Duan N, e.a., Nuclear Science and Techniques, 2013: p. 24:010301.
17. Michalet, X., et al., Dynamic molecular combing: stretching the whole human genome for high-resolution studies. Science, 1997. 277(5331): p. 1518-23.
18. Hu, J., et al., Imaging of Single Extended DNA Molecules on Flat (Aminopropyl)triethoxysilane?Mica by Atomic Force Microscopy. langmuir, 1996. 12(7): p. 1697-1700.
19. Magnello, M.E., Karl Pearson and the Origins of Modern Statistics: An Elastician becomes a Statistician. Rutherford Journal, 2005(2005-2006).